L’intelligence artificielle prédit les signatures génétiques immunitaires et inflammatoires directement à partir de l’histologie du carcinome hépatocellulaire. Une étude dirigée par le Pr Julien Calderaro et publiée dans Journal of Hepatology
 Les signatures géniques immunitaires reflétant la qualité et la quantité de l’infiltration immunitaire intra-tumorale sont connues pour prédire la sensibilité à l’immunothérapie mais leur utilisation dans un contexte clinique est cependant délicate, car elles nécessitent l’accès à des plateformes de biologie moléculaire pour l’extraction des acides nucléiques et leur traitement/séquençage. Elles dépendent également fortement de la qualité des échantillons et sont sujettes à des problèmes de normalisation et d’expertise bio-informatique.
Les signatures géniques immunitaires reflétant la qualité et la quantité de l’infiltration immunitaire intra-tumorale sont connues pour prédire la sensibilité à l’immunothérapie mais leur utilisation dans un contexte clinique est cependant délicate, car elles nécessitent l’accès à des plateformes de biologie moléculaire pour l’extraction des acides nucléiques et leur traitement/séquençage. Elles dépendent également fortement de la qualité des échantillons et sont sujettes à des problèmes de normalisation et d’expertise bio-informatique.
En revanche, les lames histologiques sont facilement disponibles dans les services de pathologie. Il est bien établi que ces lames contiennent une grande quantité d’informations qui permettent un diagnostic définitif et la prédiction du pronostic. L’avènement de la pathologie numérique et de l’intelligence artificielle (IA) permet également de standardiser leur analyse et d’extraire des caractéristiques morphologiques significatives qui ne sont pas facilement accessibles à l’œil humain.
Dans une étude preuve de concept dirigée par le Pr Julien Calderaro (Inserm U955, Equipe Pawlotsky), les auteurs ont développé et validés des modèles d’intelligence artificielle capables de caractériser, à partir des simples lames histologiques numériques, les caractéristiques immunitaires des cancers du foie.
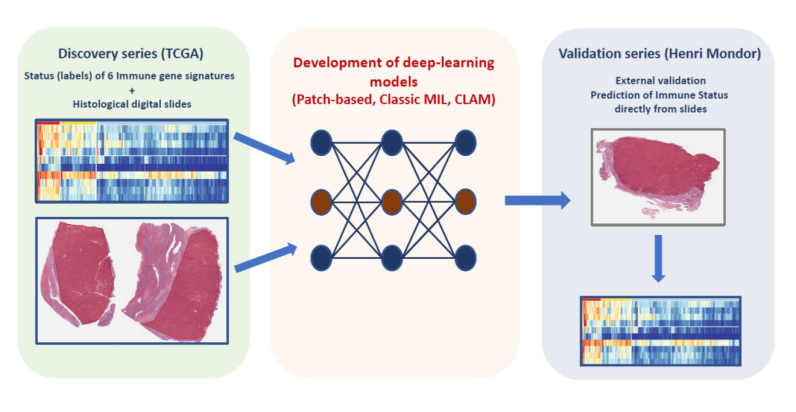
Ce type d’approche pourrait constituer un nouveau type de biomarqueur, et pourrait permettre d’identifier plus rapidement les patients pouvant bénéficier de thérapies immunomodulatrices.
Ces travaux ont été publiés dans la revue Journal of Hepatology (IF 25) du 7 février 2022 (full text via Insermbiblio)
Zeng Q, Klein C, Caruso S, Maille P, Laleh NG, Sommacale D, Laurent A, Amaddeo G, Gentien D, Rapinat A, Regnault H, Charpy C, Nguyen CT, Tournigand C, Brustia R, Pawlotsky JM, Kather JN, Maiuri MC, Loménie N, Calderaro J.

